1. Introduction▲
Le langage R, distribué gratuitement, est très utilisé dans le domaine des statistiques et de l'analyse des données, notamment grâce à une bibliothèque large et complète. Perl est un langage de script qui permet de manipuler aisément processus et fichiers textes. Les mathématiciens et bio-informaticiens l'apprécient également pour sa gestion des expressions régulières. Il peut donc être très intéressant de conjuguer ces deux langages, afin de profiter de leurs avantages respectifs.
Statistics::R (Controls the R interpreter through Perl) est l'un des modules qui permet de faire interagir les langages R et Perl.
Il est ainsi possible dans un script Perl de faire appel à R et à ses bibliothèques, de récupérer les objets ainsi créés et de les exploiter dans le pipeline d'analyse.
Les étapes principales d'utilisation du module sont les suivantes :
- déclaration de l'objet Perl $R ;
- ouverture du pont entre R et Perl ;
- chargement des bibliothèques et/ou création des objets R et/ou des calculs statistiques ;
- fermeture du pont.
On prendra ici l'exemple de l'analyse de données transcriptomiques en utilisant la bibliothèque FactoMineRFactoMineR. L'un des scripts ci-dessous pourra donc être utile aux bio-informaticiens débutants !
2. Contexte▲
L'exemple est basé sur l'étude de données transcriptomiques. Le fichier de soumission contient les données d'expression de 20 562 gènes pour deux conditions A et B, chacune représentée par quatre échantillons (de A1 à A4 et de B1 à B4).
Ce tableau de données contient donc huit échantillons (huit individus ou huit colonnes) et 20 652 gènes (variables quantitatives). Une variable qualitative sera ajoutée pour décrire chaque échantillon.
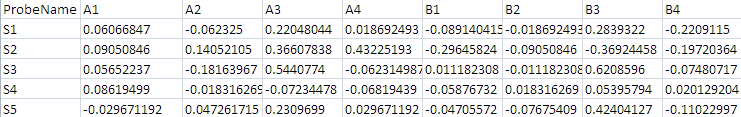
L'objectif de cette étude est de résumer et décrire ce jeu de données et d'identifier des relations avec les conditions A et B.
Classiquement, le fichier de données représente les gènes en lignes et les individus en colonnes. En effet, en transcriptomique, le nombre de gènes est très largement supérieur au nombre d'échantillons. Ce format permet ainsi d'être supporté par la plupart des tableurs et des éditeurs de texte.
Pour l'utilisation de la fonction ACP (analyse en composantes principales) disponible dans la bibliothèque FactoMineR, ce tableau sera donc importé et transposé.
3. Options et méthodes▲
Les principales options du module Statistics::R sont les suivantes :
- log_dir : espace de travail. Il représente le répertoire de travail où le pont sera créé entre les deux langages. R et Perl doivent y avoir les droits de lecture et d'écriture ;
- r_bin : chemin vers l'exécutable R.
Les méthodes principales du module Statistics::R sont les suivantes :
- startR : ouverture du pont entre R et Perl ;
- start_sharedR : ouverture du pont ou utilisation d'une communication existante ;
- stopR : fermeture du pont entre R et Perl ;
- Rbin : retour du chemin de l'exécutable R ;
- send ($CMD) : envoi des commandes à exécuter par R ;
- is_started : TRUE si l'interpréteur R est démarré ;
- clean_up : nettoyage de l'environnement en supprimant tous les objets ;
- error : retour du dernier message d'erreur.
Remarque : il est bien sûr nécessaire que R soit préalablement installé !
Pour l'envoi des commandes R, on utilise la commande qq`` dans le cas où il est nécessaire d'interpoler des variables Perl ou pour lire les objets créés par Perl :
$R->send(
qq`
`
);
Dans le cas contraire, on utilise la commande q``.
$R->send(
q`
`
);
Script pas-à-pas :
- déclaration de l'espace de travail et du chemin de l'exécutable R :
#!/usr/bin/perl
use strict;
use Carp;
use warnings;
use Statistics::R;
# Espace de travail, répertoire SPÉCIFIQUE pour l'établissement du pont entre R et Perl
my $log_dir = 'C:/Documents and Settings/stoyak/Article_Dvp/LogDir';
# Rbin path
my $r_bin = 'C:/Program Files/R/R-2.12.0/bin/R.exe';
- déclaration de l'objet $R et ouverture du pont :
# Déclaration de l'objet
my $R = Statistics::R->new(
"r_bin" => $r_bin,
"log_dir" => $log_dir,
) or die "Problem with R : $!\n";
# Ouverture du pont
$R->startR;
- création d'un objet :
# Création d'un objet
$R->send(
q`
x<-1
y<-2
sum<-x+y
`
);
- lecture de l'objet dans le script Perl:
# Lecture de l'objet
$R->send(
qq`
sum<-x+y
\n print(sum)
`
);
my $sum = $R->read;
print $sum,"\n";
- création d'une image :
# Création d'une image
my $image_pdf = 'C:/Documents and Settings/stoyak/Article_Dvp/plot.pdf';
$R->send(
qq`
pdf("$image_pdf")
v1<-c(1,2,3)
v2<-c(4,5,6)
plot(v1,v2,type="l",main="exemple de plot")
dev.off()
`
);
- chargement d'un fichier de données ("Data.txt") qui contient les informations suivantes :
| Data | Col1 | Col2 | Col3 |
| Row1 | A | 1 | z |
| Row2 | B | 2 | y |
| Row3 | C | 3 | x |
| Row4 | D | 4 | w |
# Chargement d'un fichier de données
my $file = 'C:/Documents and Settings/stoyak/Article_Dvp/Data.txt';
$R->send(
qq`
data<-read.table("$file", header=T, row.names=1, sep="\t")
\n print(data)
`
);
my $data = $R->read;
print $data,"\n\n";
- chargement d'une bibliothèque :
# Chargement d'une bibliothèque
$R->send(
q`
library(FactoMineR)
`
);
- sauvegarde des objets :
# Sauvegarde des objets
my $r_save = 'C:/Documents and Settings/stoyak/Article_Dvp/Save.RData';
$R->send(
qq`
save(v1,v2,data, file="$r_save")
`
);
- suppression des objets :
# Suppression des objets
$R->send(
qq`
rm(v1,v2,data)
ls<-ls()
\n print(ls)
`
);
my $ls = $R->read;
print $ls,"\n\n";
- chargement du fichier .Rdata
# Chargement du fichier .RData
$R->send(
qq`
load("$r_save")
ls<-ls()
\n print(ls)
`
);
# Vérification du chargement
my $load = $R->read;
print $load,"\n\n";
$R->send(
qq`
\n print(v1)
`
);
my $v1 = $R->read;
print $v1,"\n\n";
- fermeture du pont :
# Fermeture du pont
$R->stopR();
Et voici le script de pas-à-pas complet :
#!/usr/bin/perl
use strict;
use Carp;
use warnings;
use Statistics::R;
# Espace de travail, répertoire SPÉCIFIQUE pour l'établissement du pont entre R et Perl
my $log_dir = 'C:/Documents and Settings/stoyak/Article_Dvp/LogDir';
# Rbin path
my $r_bin = 'C:/Program Files/R/R-2.12.0/bin/R.exe';
# Déclaration de l'objet $R
my $R = Statistics::R->new(
"r_bin" => $r_bin,
"log_dir" => $log_dir,
) or die "Problem with R : $!\n";
# Ouverture du pont
$R->startR;
# Création d'un objet
$R->send(
q`
x<-10
y<-2
`
);
# Lecture de l'objet
$R->send(
qq`
sum<-x+y
\n print(sum)
`
);
my $sum = $R->read;
print $sum,"\n\n";
# Création d'une image
my $image_pdf = 'C:/Documents and Settings/stoyak/Article_Dvp/plot.pdf';
$R->send(
qq`
pdf("$image_pdf")
v1<-c(1,2,3)
v2<-c(4,5,6)
plot(v1,v2,type="l",main="exemple de plot")
dev.off()
`
);
# Chargement d'un fichier de données
my $file = 'C:/Documents and Settings/stoyak/Article_Dvp/Data.txt';
$R->send(
qq`
data<-read.table("$file", header=T, row.names=1, sep="\t")
\n print(data)
`
);
my $data = $R->read;
print $data,"\n\n";
# Chargement d'une bibliothèque
$R->send(
q`
library(FactoMineR)
`
);
# Sauvegarde des objets
my $r_save = 'C:/Documents and Settings/stoyak/Article_Dvp/Save.RData';
$R->send(
qq`
save(v1,v2,data, file="$r_save")
`
);
# Suppression des objets
$R->send(
qq`
rm(v1,v2,data)
ls<-ls()
\n print(ls)
`
);
my $ls = $R->read;
print $ls,"\n\n";
# Chargement du fichier .RData
$R->send(
qq`
load("$r_save")
ls<-ls()
\n print(ls)
`
);
# Vérification du chargement
my $load = $R->read;
print $load,"\n\n";
$R->send(
qq`
\n print(v1)
`
);
my $v1 = $R->read;
print $v1,"\n\n";
# Fermeture du pont
$R->stopR();
À vos tests !!
4. Exemples▲
4-A. Exemple de données transcriptomiques▲
Pour les bio-informaticiens, cet exemple consiste en l'étude de données transcriptomiques, en utilisant la méthode ACP de la bibliothèque FactoMineR.
La procédure ACP ci-dessous consiste en ces quelques étapes :
- déclaration des variables Perl et de l'objet Statistics::R ;
- ouverture du pont entre R et Perl ;
- chargement de la bibliothèque FactoMineR ;
- import et transposition du fichier de données ;
- ajout de la variable qualitative « condition » ;
- appel de la fonction PCA et création du fichier avec les graphes sur les variables et les individus ;
- calcul du pourcentage d'inertie pour les quatre premiers axes ;
- description des axes ;
- lecture des objets créés par R dans Perl ;
- sauvegarde des objets dans un fichier .Rdata ;
- fermeture du pont entre R et Perl.
Voici le script complet :
#!/usr/bin/perl
use strict;
use Carp;
use warnings;
use File::Basename;
use Statistics::R;
#===========================================
# Auteur : Stoyak - Developpez.com
# But : Statistics-R demo
# Date : 27/07/2011 17:30:00
#===========================================
#===========================================
# Définition des paramètres
#===========================================
# Fichier de données
my $file = 'C:/Documents and Settings/stoyak/Article_Interaction/Data.txt';
# Espace de travail, répertoire SPÉCIFIQUE pour l'établissement du pont entre R et Perl
my $log_dir = 'C:/Documents and Settings/stoyak/Article_Interaction/LogDir';
# Rbin path
my $r_bin = 'C:/Program Files/R/R-2.12.0/bin/R.exe';
my %parameters = (
'file' => $file,
'log_dir' => $log_dir,
'r_bin' => $r_bin,
);
#===========================================
# Appel de la procédure ACP
#===========================================
ACP( \%parameters );
#===========================================
# Procédure ACP
#===========================================
sub ACP {
my ( $ref_params ) = @_;
my ( $file_name, $dir_name, $extension ) = fileparse( $ref_params->{file}, qr/\.[^.]*/ );
# Fichiers de résultats ('.txt' et '.pdf')
my $pdf_acp = $dir_name . '/PCA.pdf';
my $result_file = $dir_name . '/Result.txt';
open my $fh, '>', $result_file || die "Impossible d'écrire dans le fichier $result_file\n";
# Déclaration de l'objet
my $R = Statistics::R->new(
"r_bin" => $ref_params->{r_bin},
"log_dir" => $ref_params->{log_dir},
) or die "Problem with R : $!\n";
# Ouverture du pont
$R->startR;
# Chargement de la bibliothèque FactoMineR (http://factominer.free.fr/)
# q` pour le chargement de la bibliothèque
$R->send(
q`
library(FactoMineR)
`
);
# Transposition du fichier original
# qq` pour l'interpolation des variables Perl
$R->send(
qq`
data<-read.table("$ref_params->{file}", header=T, row.names=1, sep="\t")
data<-as.data.frame(t(data))
condition<-as.factor(c(rep("A",4),rep("B",4)))
data<-cbind.data.frame(condition,data)
colnames(data)[1]<-"condition"
`
);
# Fichier avec les graphes sur les variables et les individus
$R->send(
qq`
res.pca<-PCA(data,ncp=6,graph=FALSE,quali.sup=1)
pdf("$pdf_acp")
plot(res.pca, choix="ind", axes=1:2, new.plot=F)
plot(res.pca, choix="var", axes=1:2, new.plot=F)
plot(res.pca, choix="ind", axes=2:3, new.plot=F)
plot(res.pca, choix="var", axes=2:3, new.plot=F)
dev.off()
`
);
# Pourcentage d'inertie pour les quatre premiers axes
# qq` pour la récupération de l'objet round par Perl
$R->send(
qq`
round<-round(res.pca\$eig[1:4,],2)
\n print(round)
`
);
my $inertie = $R->read;
# Description des axes
$R->send(
qq`
dimdesc<-dimdesc(res.pca)
\n print(dimdesc)
`
);
my $dimdesc = $R->read;
# Affichage des objets "round" et "dimdesc" dans le fichier de résultat
print $fh "Inertia:\n$inertie\n\nDimdesc:\n$dimdesc\n";
# Sauvegarde des objets "data'" et "res.pca" (fichier .RData)
my $r_save = $dir_name . '/Data.RData';
$R->send(
qq`
save(data,res.pca, file="$r_save")
`
);
# Fermeture du pont
$R->stopR();
close $fh;
return;
}
On obtient donc trois fichiers :
- "Data.RData"
- "PCA.pdf" (exemple du graphe des individus selon les deux premiers axes)
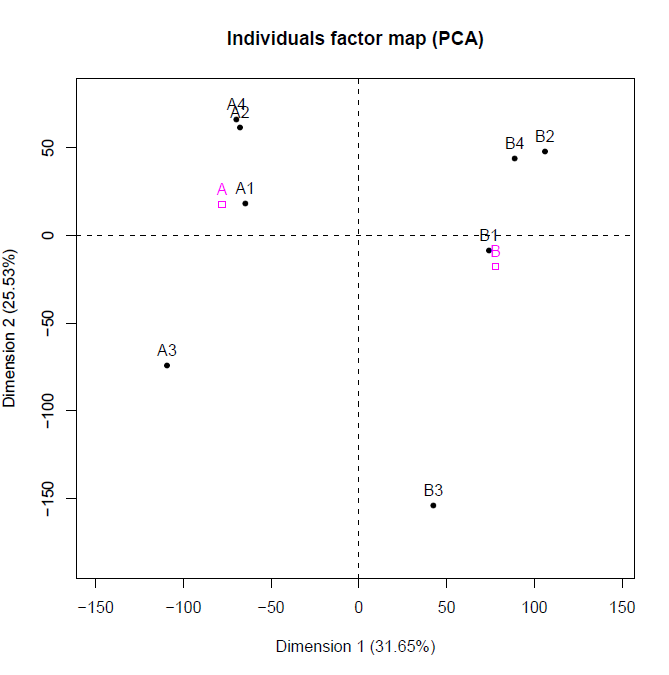
- "Result.txt" (extrait du fichier de résultat)
Inertia:
eigenvalue percentage of variance cumulative percentage of variance
comp 1 6508.60 31.65 31.65
comp 2 5249.58 25.53 57.18
comp 3 2841.99 13.82 71.01
comp 4 2036.85 9.91 80.91
Dimdesc:
$Dim.1
$Dim.1$quanti
correlation P-value
S213 0.9975904 3.491288e-08
S7134 0.9957727 1.882513e-07
S9211 0.9954746 2.309098e-07
S15923 0.9937002 6.221021e-07
S4168 0.9931895 7.857080e-07
.../...
$Dim.1$quali
P-value
condition 9.77511e-05
$Dim.1$category
Estimate P-value
B 77.91396 9.77511e-05
A -77.91396 9.77511e-05
$Dim.2
$Dim.2$quanti
correlation P-value
S10368 0.9911161 1.741237e-06
S6443 0.9910109 1.803658e-06
S5688 0.9907445 1.968439e-06
S442 0.9900412 2.450838e-06
S1041 0.9892404 3.089000e-06
.../...
$Dim.3
$Dim.3$quanti
correlation P-value
S6447 0.9858095 7.068008e-06
S12141 0.9729702 4.837528e-05
S16799 0.9705853 6.223013e-05
S193 0.9672014 8.605207e-05
S17531 0.9661212 9.475946e-05
S11034 0.9645758 1.082002e-04
.../...
4-B. Exemple de la bibliothèque FactoMineR▲
Pour les non-bio-informaticiens, ou pour ceux qui souhaiteraient tester ces méthodes sans avoir de fichier à disposition, un exemple est disponible dans la bibliothèque FactoMineR. Le jeu de données consiste en un tableau qui contient les performances réalisées par des athlètes lors de deux compétitions. Le fichier est disponible. Le script présenté ci-dessous reprend les codesles codes qui vous sont détaillés par les auteurs de la bibliothèque.
Les données :
Le tableau de données contient 41 lignes et 13 colonnes.
Les colonnes 1 à 12 sont des variables continues: les dix premières colonnes correspondent aux performances des athlètes pour les dix épreuves du décathlon et les colonnes 11 et 12 correspondent respectivement au rang et au nombre de points obtenus. La dernière colonne est une variable qualitative correspondant au nom de la compétition (Jeux Olympiques de 2004 ou Décastar 2004).
Et voici donc le script !
#!/usr/bin/perl
use strict;
use Carp;
use warnings;
use File::Basename;
use Statistics::R;
#===========================================
# Auteur : Stoyak - Developpez.com
# But : Statistics-R demo
# Date : 27/07/2011 21:30:04
#===========================================
#===========================================
# Définition des paramètres
#===========================================
# Répertoire de sortie
my $dir = 'C:/Documents and Settings/stoyak/Article_Dvp';
# Espace de travail, répertoire SPÉCIFIQUE pour l'établissement du pont entre R et Perl
my $log_dir = 'C:/Documents and Settings/stoyak/Article_Dvp/LogDir';
# Rbin path
my $r_bin = 'C:/Program Files/R/R-2.12.0/bin/R.exe';
my %parameters = (
'dir' => $dir,
'log_dir' => $log_dir,
'r_bin' => $r_bin,
);
#===========================================
# Appel de la procédure ACP
#===========================================
ACP( \%parameters );
#===========================================
# Procédure ACP
#===========================================
sub ACP {
my ( $ref_params ) = @_;
# Fichiers de résultats ('.txt' et '.pdf')
my $pdf_acp = $ref_params->{dir} . '/DecathPCA.pdf';
my $result_file = $ref_params->{dir} . '/DecathResult.txt';
open my $fh, '>', $result_file || die "Impossible d'écrire dans le fichier $result_file\n";
# Déclaration de l'objet
my $R = Statistics::R->new(
"r_bin" => $ref_params->{r_bin},
"log_dir" => $ref_params->{log_dir},
) or die "Problem with R : $!\n";
# Ouverture du pont
$R->startR;
# Chargement de la bibliothèque FactoMineR (http://factominer.free.fr/) et des données
$R->send(
q`
library(FactoMineR)
data(decathlon)
`
);
# ACP sur les individus et les variables
$R->send(
qq`
pdf("$pdf_acp")
res.pca<-PCA(decathlon, scale.unit=TRUE, ncp=5, quanti.sup=c(11: 12), quali.sup=13, graph=FALSE)
plot(res.pca, choix="ind", axes=1:2, new.plot=F, habillage=13)
plot(res.pca, choix="var", axes=1:2, new.plot=F, habillage=13)
dev.off()
`
);
# Description des axes
$R->send(
qq`
dimdesc<-dimdesc(res.pca, axes=c(1,2))
\n print(dimdesc)
`
);
my $dimdesc = $R->read;
# Print de l'objet 'dimdesc' dans le fichier de résultat
print $fh "Dimdesc:\n$dimdesc\n";
# Fermeture du pont
$R->stopR();
close $fh;
return;
}
Après exécution, vous devez avoir dans votre répertoire de sortie les deux fichiers "DecathPCA.pdf" et "DecathResult.txt" dont les contenus sont décrits par les auteurs.
5. Remerciements▲
Je tiens à remercier djibril de m'avoir suggéré d'écrire cet article et de m'avoir introduit dans la communauté des rédacteurs de developpez.com.
Je tiens également à remercier _Max_, ClaudeLELOUP et dourouc05 pour leurs corrections.
Je tiens à citer de nouveau la bibliothèque FactoMineR grâce à laquelle j'ai exposé des exemples plus complexes et complets.





